实验室计算生物团队新突破:通过侧链建模研究蛋白质突变的影响及序列改造
来源:上海人工智能实验室 | 2023-02-27
近日,上海人工智能实验室联合复旦大学提出蛋白质点突变建模新方法——通过侧链建模研究蛋白质突变的影响及序列改造。该方法由上海人工智能实验室计算生物学创新中心教授、复旦大学复杂体系多尺度研究院院长马剑鹏和诺贝尔化学奖得主Michael Levitt教授共同带领的科研团队提出。团队从蛋白质侧链结构出发对点突变问题进行深入探索,并利用早前开发的新算法(OPUS-Mut)指导蛋白质设计,为研究蛋白质突变和蛋白质设计问题提供了新的利器。目前,该成果已开源,相关论文“OPUS-Mut: Studying the Effect of Protein Mutatio through Side-Chain Modeling”在线发表于知名国际期刊Journal of Chemical Theory and Computation。
蛋白质点突变在蛋白质工程和蛋白质进化的研究中起重要作用,也是诸多重大遗传疾病的发病原因,因此,对点突变的结构进行建模意义重大。然而,目前最优的蛋白质结构预测算法AlphaFold2并不适合直接应用于点突变建模[1],这使得高精度点突变结构建模成为全球计算生物界普遍面临的难题。此次团队提出的通过侧链建模研究蛋白质突变影响的方法,正是针对这一世界难题做出的突破性研究。
从结构角度来说,蛋白质点突变可以近似地看作是相应残基的侧链替换。因此,从侧链结构出发对点突变问题进行研究是一条可行之路。经过算法改进和模型设计,团队所开发的算法对突变位点侧链构象的预测有着较高的灵敏度(图1)。同时,通过对比野生蛋白和突变蛋白的侧链建模结果,可以得到由突变引起的、对蛋白中其它残基的侧链结构扰动数据,进而预测该突变对蛋白功能的影响。研究团队收集了293个T4溶菌酶点突变对稳定性的影响数据,并将其分为提升稳定性组与降低稳定性组。结果显示(图2),两组的突变对突变前后其他残基侧链扰动的大小存在显著性差异,且降低稳定性组的突变对其他残基扰动更大——在293个突变中,扰动最大的前10个突变中有9个属于降低稳定性组。与此同时,该篇论文中的其他实验也得出了类似结果,这些实验共同验证了新算法可以用来区分“温和型突变”和“破坏型突变”,即对目标蛋白中其他残基的侧链结构扰动越大,则该突变更可能为“破坏型突变”。由于该算法并未使用任何突变-生物功能变化数据进行训练,故不存在过拟合现象,可以和相关模型形成良好互补。

图1. 新算法对野生型肌红蛋白及其突变体在突变位点的侧链建模结果。a)为突变位点的周围环境。b-f)分别为野生型肌红蛋白和其不同突变体的侧链建模结果。其中红色为突变位点真实实验结构,蓝色为突变位点周围残基的真实实验结构,黄色为预测结果。

图2. 提升稳定性组和降低稳定性组的侧链扰动大小(Sdiff)。显著性分析采用Mann-Whitney U-test。
此外,研究团队以T4溶菌酶为目标蛋白,遍历其所有可能的点突变(164×19),之后计算不同点突变对其它残基侧链的扰动情况,并对其按照扰动大小进行排序,然后选用对其它残基侧链扰动最小的那部分点突变进行组合,得到了一个具有60%序列相似度的T4溶菌酶的突变体(图3)。同时,在不采用模板信息的情况下使用AlphaFold2对该突变体进行结构预测,预测结果与原始野生型蛋白的真实实验结构的相似度指标TM-score为0.95。该结果显示,新算法可以在降低序列同源性的同时,保持目标蛋白的生物学功能,从而丰富具有相同功能的蛋白序列多样性,助力蛋白质设计。值得注意的是,由于其并未用到多序列比对信息,故可以和基于多序列比对及序列采样信息的蛋白质幻想设计方法形成良好互补,对其采样提供一定指导。
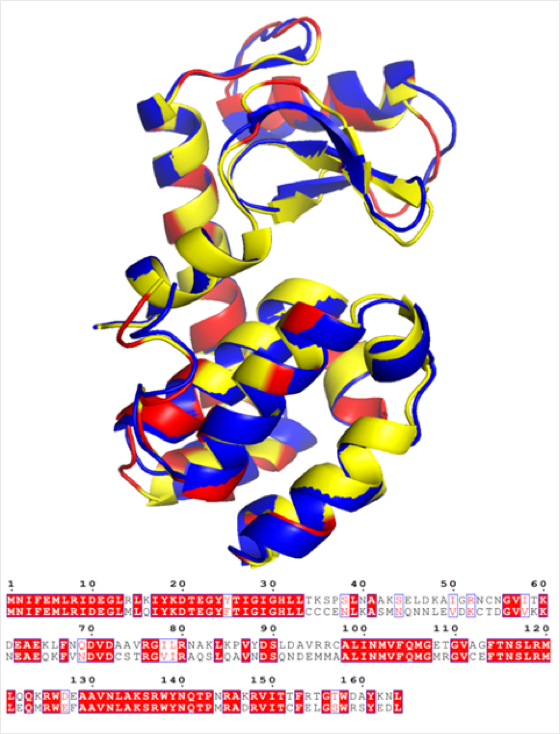
图3. 具有60%序列相似度的T4溶菌酶突变体的AlphaFold2预测结果。蓝色为原始野生型蛋白的真实实验结构,黄色为AlphaFold2对突变体的预测结构,红色为突变位点。
马剑鹏教授与Michael Levitt教授的联合团队在蛋白质结构预测和设计领域深耕已久,科研成果发表于Nature、Science、PNAS等国际顶刊。近年来,团队相继开发了OPUS-Fold、OPUS-Refine、OPUS-X、OPUS-Rota4及OPUS-Mut等一系列算法,可分别应用于不同场景,为蛋白质结构预测和设计提供了坚实的技术保障。尤其是对基于蛋白质大分子药物的设计与优化,团队具有干湿结合、AI赋能的全链条一体化的研发优势。
上海人工智能实验室计算生物学创新中心以人工智能为技术中枢,构建新一代生物体系分析工具与方法,解读生物遗传信息,加速生命科学在分子层面的研究。通过对蛋白质、DNA等分子功能结构的预测与设计,打破生物数据壁垒,为生命科学研究提供新思路、新方法。
[1] G. R. Buel, K. J. Walters, Can AlphaFold2 predict the impact of missense mutations on structure? Nat Struct Mol Biol29, 1-2 (2022).


